Genomas, diversidad y variación natural
En la naturaleza, la información genética es increíblemente dinámica y fundamental para comprender la diversidad y la evolución de la vida en nuestro planeta. En este artículo realizaremos un viaje por la célula y revisaremos qué es el ADN y sus niveles de complejidad, hasta llegar a los genes y los genomas. Veremos cómo en los seres humanos, nuestro ADN se organiza en 23 pares de cromosomas, sumando un total de 46. Sin embargo, el mundo natural es increíblemente diverso: la papa mexicana es tetraploide, con juegos de 4pares de cromosomas, mientras que en las suculentas mexicanas del género Echeveria, algunas especies tienen entre 12 y 135 pares de cromosomas, y el helecho Ophioglossum ostenta el récord con 630 pares. Esto muestra cómo el genoma puede variar significativamente entre especies. Además, entenderemos que las mutaciones, o cambios en el código genético, pueden ocurrir en diferentes magnitudes y formas: desde la replicación del genoma, la recombinación genética, hasta la actividad de elementos saltarines o transposones.
Del ADN al genoma
El genoma es el acervo de información que contiene las instrucciones para formar un organismo, mantener todos sus componentes y cómo estos interactúan con el ambiente a lo largo de su desarrollo. El genoma, en su esencia, es el texto de la vida, contenido en el ADN, y está escrito en un lenguaje de ácidos nucleicos, que, por su estructura química, también se les denomina bases nitrogenadas: adenina (A), timina (T), guanina (G) y citosina (C). El ADN es una doble cadena y cada una de estas bases nitrogenadas tiene su par complementario, de tal manera que una cadena de ATGC tiene su cadena complementaria TACG. Es por eso que cuando nos referimos al código genético empleamos la unidad “pares de bases”, que se refiere al ADN y códigos de información contenidos en ambas cadenas complementarias (Fig. 1). Por el número de letras, pareciera un lenguaje muy rudimentario comparado con el alfabeto español, pero con solo cuatro caracteres (A, T, G y C) con el que podríamos formar 256 palabras de cuatro letras: ATGC, AGTC, AATGG, TGCA, etc (Hayes et al., 1998)
Cada una de las células contiene el genoma del organismo. Es decir, en nuestro cuerpo, el genoma de las células de la piel es el mismo que el de las neuronas, de los glóbulos blancos, etc. Dentro de la célula, el genoma está empaquetado en fascículos, llamados cromosomas. Al conjunto de todos los cromosomas presentes en la célula se le llama genoma. En los animales, como en los humanos, parte del genoma se encuentra tanto en el núcleo como en las mitocondrias. En plantas, el genoma también se encuentra en un organelo encargado de la fotosíntesis, llamado cloroplasto. La porción del genoma que contienen las mitocondrias y los cloroplastos es proporcionalmente mucho menor que el tamaño del genoma del núcleo. El genoma de un cloroplasto ronda entre 110 y 200 mil pares de bases, mientras que el de la mitocondria puede ser de 11 a 28 mil pares de bases (Cui et al., 2006; Formaggioni et al., 2021). En comparación, el cromosoma 1 es el más grande del ser humano y tiene 247 millones de pares de bases, mientras que el más pequeño es más de cinco veces menor y tiene cerca de 47 millones de pares de bases (Erickson & Franciszkowicz,2010).
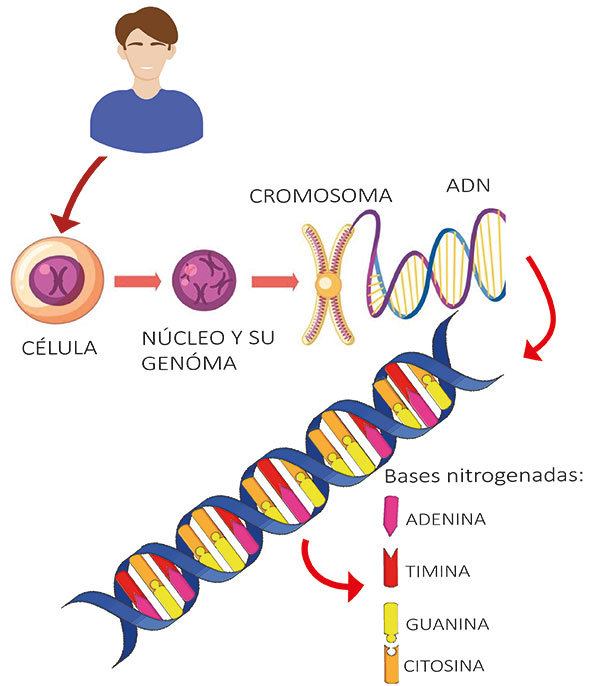
Fig. 1. En cada una de las células con núcleo se encuentra el genoma empaquetado en forma de cromosomas. Cada cromosoma es un fragmento de ADN ultracompactado. A su vez el ADN está conformado por cuatro bases: Adenina, Timina, Citosina y Guanina.
En la realidad, los genomas no tienen solo cuatro pares de bases, sino que varían mucho en tamaño. El genoma más pequeño que se conoce es el de una bacteria simbionte de insectos, llamada Candidatus Hodgkinia, cuyo genoma tiene 144 mil pares de bases, lo cual no es nada despreciable. Aunque es muy pequeño si lo comparamos con el genoma más grande conocido, que pertenece a una planta de la familia Melanthiaceae, Paris japonica, con 150,000 millones de pares de bases, que, si lo entendiéramos linealmente, tendría una extensión de aproximadamente 90 metros. Esto también significa que es casi un millón de veces más grande que el genoma de Candidatus Hodgkinia, lo que evidencia el amplio espectro de tamaños que pueden presentar los genomas. Para ponernos en contexto, el genoma humano tiene 3,200 millones de pares de bases, el agave tequilero (Agave tequilana) tiene 7,500 millones, el maíz (Zea mays) tiene 2,300 millones, mientras que el genoma del arroz (Oryza sativa) es más pequeño que el del humano, con 420 millones de pares de bases (Purugganan & Jackson,2021) (Fig. 2). Es decir, no hay relación directa entre el tipo de organismo y el tamaño de su genoma, sino que el tamaño del genoma es un atributo de las especies que evoluciona de manera espectacularmente dinámica, tanto entre especies lejanamente relacionadas, como entre poblaciones de la misma especie. Cabe preguntarse cómo se origina tal diversidad de tamaños del genoma. La respuesta puede estar en entender cómo se empaqueta el genoma en cada una de las células de un organismo.
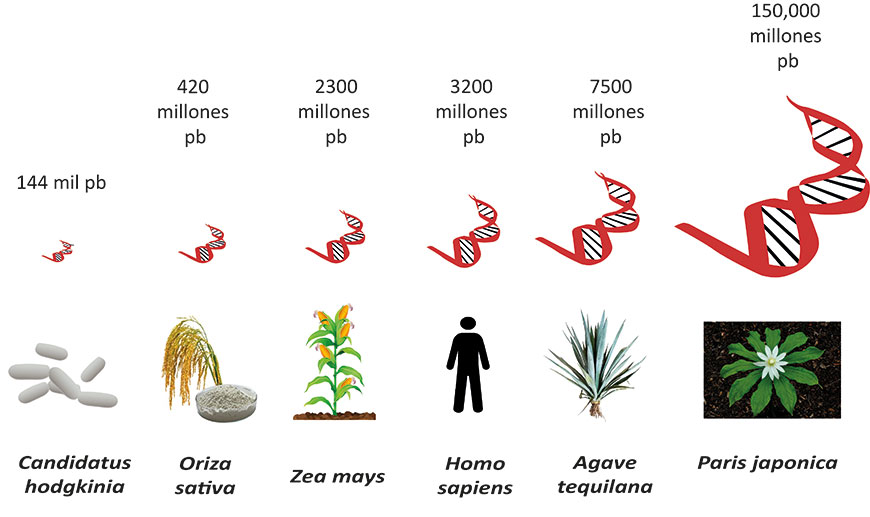
Fig. 2. Diversidad de tamaños de los genomas de distintos seres vivos, desde bacterias hasta plantas.
En organismos como los humanos, el genoma nuclear está dispuesto en pares de cromosomas, de modo que tenemos 23 pares de fascículos nucleares, es decir, 46 en total, y por lo tanto somos organismos diploides. En otros organismos, es frecuente tener 2 o mas pares de cromosomas, por ejemplo, la papa mexicana Solanum tuberosum es tetraploide (4 pares de cromosomas), los peces ciegos Astyanax spp. pueden ser diploides o triploides (3 pares de cromosomas), mientras que las especies de nopales pueden ser desde diploides, hasta octaploides (ocho pares de cromosomas); pero el récord actualmente lo tiene una especie de helecho llamada Ophioglossum que tiene 630 pares de cromosomas en cada célula (CA, 1958). Es decir, el genoma alojado en el núcleo es altamente dinámico en términos del tamaño de cada uno de los cromosomas, y en número de juegos de cromosomas que pueden tener las especies.
En el Instituto de Biología, estudiamos estos fenómenos que ocurren a nivel de la arquitectura del genoma para entender cómo han contribuido a la evolución de las especies. Tenemos ejemplos de la evolución del genoma en especies suculentas mexicanas, como los nopales, donde organismos de la misma especie pueden variar en el tamaño del genoma en 2000 millones de pares de bases (casi un genoma humano); o en las siemprevivas de la familia Crassulaceae del género Echeveria, y en las que hemos visto que hay especies con 12 pares de cromosomas (Echeveria catorce), hasta especies con 135 pares de cromosomas (Echeveria aff. fulgens) (Palomino et al., 2021).
Genes en acción: los ingenieros escondidos de la vida
Es a partir de la gran cantidad de información escrita en el lenguaje de pares de bases que se conforman los genes, que son paquetes de información que llevan instrucciones para que se realice alguna función en el organismo. El tipo de genes que mejor entendemos en la actualidad son los que contienen instrucciones para la síntesis de proteínas. Las proteínas son biomoléculas que constituyen aproximadamente el 20% de nuestro cuerpo y el 80% del tejido muscular en los seres humanos. A los genes que contienen instrucciones para la síntesis de proteínas les llamamos genes codificantes. En el genoma humano existen más de 20,000 genes codificantes; en el maíz hay más de 45,000; en el musgo Physcomitrella patens, que es una planta no vascular, hay 28,000, y el álamo (Poplar sp.) tiene cerca de 42,000 (Haberer et al., 2005; NCBI, 2025; Liu et al., 2016)).Es decir, la densidad de genes codificantes de proteínas es altamente diversa entre las especies y no tiene relación directa con el tamaño del genoma: puede haber genomas pequeños con alta densidad de genes codificantes de proteínas, o genomas muy grandes con baja densidad de genes codificantes.
Las mutaciones como motores de la diversidad biológica
Toda la gran diversidad genómica que existe se ha originado a partir de varios procesos que producen cambios en la información contenida en el código de las pares de bases, a los que llamamos mutaciones. Hablando de las mutaciones del código genético (sin hacer referencia a los cambios morfológicos que puedan ocasionar), estas pueden ser de gran magnitud, afectando cromosomas o genomas enteros, o de mediana magnitud, influenciando cientos o miles de pares de bases, o ser relativamente pequeñas, impactando un par de bases o más. Las mutaciones de gran magnitud ocurren durante la replicación del genoma o la generación de gametos (por ejemplo, los espermatozoides y el óvulo en los humanos), donde puede haber duplicación excesiva de uno o de todos los cromosomas, o puede que, al formarse los gametos, no haya habido una partición equitativa del material genético. La duplicación de todo un juego cromosómico conlleva lo que llamamos poliploidías, y la falta o el exceso de algún cromosoma lo llamamos aneuploidías (Fig. 3). En los humanos, la gran mayoría de estos eventos causan letalidad en el embrión, por lo que son poco frecuentes. En plantas, por ejemplo, la duplicación del genoma es más frecuente, y se sabe que la evolución de la gran diversidad de las plantas con flor ha estado ligada a la expansión del genoma por poliploidías y aneuploidías, y que sigue sucediendo en especies naturales. Esto explica el amplio rango de tamaño de genomas vegetales que va desde 82 millones de pares de bases en Utricularia gibba, la planta con el genoma más pequeño conocido, hasta la ya mencionada Paris japonica con 150,000 millones de pares de bases (Pellicer et al., 2010).
Algunas mutaciones de mediana magnitud también pueden ocurrir durante la generación de gametos, mediante un fenómeno conocido como recombinación genética, en el que se realiza un barajeo de los cromosomas parentales. Algunas cartas de la baraja se pueden perder, o se insertan de más, o se insertan al revés, ocasionando que el genoma pierda o gane algunos cientos o miles de pares de bases, o que las secuencias estén invertidas. En otras ocasiones, el genoma puede modificarse por la actividad de elementos del propio genoma, llamados transposones. Los transposones son fragmentos móviles del genoma, que pueden tener varios cientos de pares de bases, que saltan de un lugar a otro, a veces interrumpiendo o modificando el mensaje de la secuencia receptora, o llevándose secuencias al saltar nuevamente a otro lugar, dejando su rastro genético a lo largo del genoma. Este mecanismo puede generar que genes y familias de genes se dupliquen al viajar con ellos, que crezcan en número, que el genoma se expanda o se contraiga, que se originen nuevas funciones con genes quimera, y que esto dé origen a radiaciones evolutivas. Tal es el caso de lo que ocurrió con los insectos, de los cuales sabemos que un componente importante de su evolución genómica y diversificación está fuertemente asociada a la actividad de transposones. En plantas el ejemplo clásico de la actividad de los transposones es en los granos de maíz, donde en mazorcas de granos blancos, ocasionalmente tienen granos rojos a púrpuras (Fig. 3) (McClintock, 1948). Esto es porque en esos granos pigmentados había un transposón que obstruía el gen para sintetizar antocianinas, pero que adquirió funcionalidad al momento en que el transposón saltó a otro lugar del genoma, permitiendo que el grano, o algunos parches del grano, se pigmentaran. En este y muchos casos relacionados con transposones, el ambiente, y no necesariamente uno adverso, es el que activa la maquinaria mutagénica que los organismos, como las plantas y los animales, llevan dentro.
Finalmente, las mutaciones de pequeña magnitud son aquellas que cambian una o más letras del código por otras (Fig. 3). Comúnmente se les llama mutaciones puntuales porque sólo afectan unas pares de bases. Estas mutaciones son causadas por el ambiente (por ejemplo el, exceso de radiación solar) u ocurren de manera natural en nuestras células. Cuando comparamos organismos de la misma especie, en el genoma podemos observar una gran cantidad de variaciones genéticas de este tipo. Por ejemplo, en los humanos sabemos que cuando comparamos individuos, en promedio hay un cambio del código genético cada 300 pares de bases. Es decir, si solo tomamos en cuenta este tipo de polimorfismos, cada individuo es distinto del otro en 10 millones de sitios del genoma. Entonces cabe preguntar: ¿por qué y para qué existe tanta variación? Si somos tan distintos, ¿por qué somos tan similares? ¿Qué importancia tienen esas mutaciones en nuestro genoma?
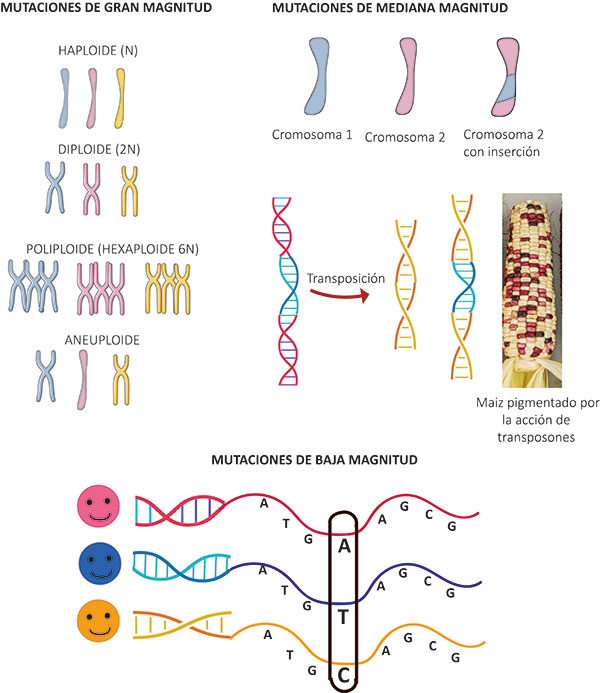
Fig. 3. Tipos de mutaciones que ocurren en los seres vivos: de baja, mediana y alta magnitud. En las mutaciones de alta magnitud, puede duplicarse uno o todos los cromosomas. En la imagen se muestra cómo sería una célula haploide con un solo juego de cromosomas, una diploide con dos juegos de cromosomas, una poliploide con tres juegos de cromosomas, mientras que en la aneuploidía se indica la pérdida de un cromosoma. En las mutaciones de mediana magnitud se observa la inserción de un fragmento de cromosoma en otro. Además, se ilustra un evento de transposición que provoca cambios en la coloración del maíz. Finalmente, en las mutaciones de baja intensidad se observan cambios de una sola base en el ADN de tres individuos.
Como mencionamos anteriormente, la magnitud de la mutación es independiente del efecto que pueda tener en los aspectos morfológicos o fisiológicos del organismo. Un ejemplo de una mutación de gran magnitud genómica es la variación en los tamaños del genoma de poblaciones de nopales que son de la misma especie, y morfológicamente son prácticamente idénticas, aunque hay una diferencia en tamaño de 2 millones de pares de bases de población a población. En contraste, mutaciones puntuales o de algunas pares de bases, como sucede en el gen CYCLOIDEA de la flor del perrito (Antirrhinum majus), pueden generar cambios drásticos en la morfología del organismo, y la flor pasa de ser bilateral a ser parcialmente radial (Clark & Coen, 2002) (Fig. 4A).
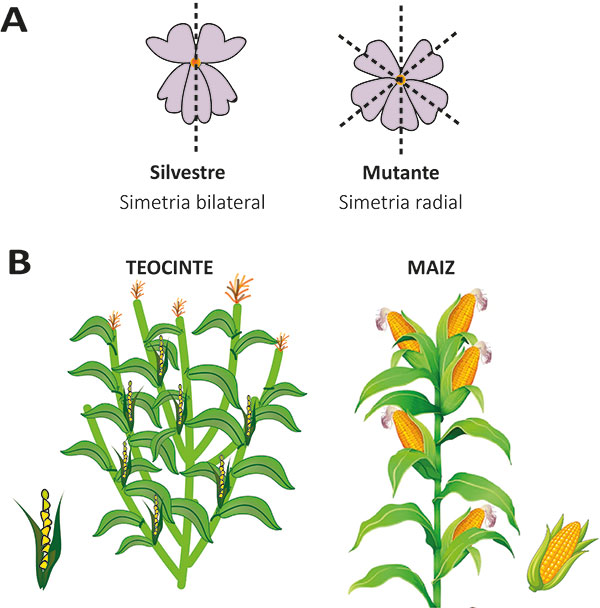
Fig. 4. A. Comparación de la planta silvestre de Antirrhinum majus con simetría lateral y de una mutante en el gen CYCLOIDEA, que origina una simetría radial. B. El teocinte es el antepasado del maíz actual y muestra una mayor ramificación que el maíz moderno, que presenta inserciones de transposones en un gen llamado TEOCINTE BRANCHED (TB1), lo que le confiere una dominancia apical sin ramificaciones.
Parte de la tarea diaria de los biólogos es entender por qué hay mutaciones que producen cambios morfológicos o fisiológicos tan drásticos, mientras que otras no. Algo que sabemos desde hace décadas es que los genes que contienen información para la síntesis de proteínas (genes codificantes) tienden a presentar menos variación que las secuencias de ADN que no codifican proteínas. Esto tiene sentido porque si la información que codifica para, por ejemplo, el colágeno, que constituye el 7% de nuestra masa corporal, por ser parte esencial de huesos, músculos y piel, tuviese mutaciones que no permitieran la correcta síntesis de la proteína, entonces nuestro organismo estaría en serios problemas. En contraparte, hay proteínas que no necesitamos de forma obligatoria. Uno de los retos para los biólogos del desarrollo es determinar cómo y cuáles mutaciones en genes codificantes generan cambios morfológicos y fisiológicos. Este es el caso de una proteína del agave tequilero, la MAYEHUELINA, que se aisló en el Instituto de Biología, donde nuestros investigadores están caracterizando su funcionamiento y su evolución para conferir tolerancia al calor y a la sequía (Lledías et al., 2020).
A lo largo de nuestra vida acumularemos cientos o miles de mutaciones en los genomas de nuestras células. Probablemente, en la edad adulta, el genoma de las células de la piel ya no sea idéntico al de las células musculares, como ocurría en un principio, cuando éramos embriones. De cualquier forma, la inmensa gran mayoría de estas mutaciones no pasarán a nuestra descendencia y, por tanto, no formarán parte del proceso evolutivo, a menos que estas mutaciones sucedan en las células germinales que dan origen a los gametos, o en los gametos mismos, que en humanos son los espermatozoides y los óvulos. Cualquier otra mutación, que no se origina en células germinales o los gametos, es llamada somática y no tiene repercusiones en la siguiente generación, pues no es heredable.
Recapitulando, una mutación es una alteración en el mensaje del código genético. Independientemente de su magnitud genética, puede o no tener efectos morfológicos o fisiológicos. Las mutaciones se generan constantemente por procesos naturales o por estímulos ambientales. Solo aquellas que se dan en células germinales o en gametos son heredables, y por tanto son parte del proceso evolutivo. La mayor parte de las mutaciones en un organismo producen efectos nulos o casi nulos en el organismo, por lo que podemos leerlas al decodificar el genoma de individuos de la misma especie. Otras mutaciones no están en el registro del genoma porque han sido seleccionadas o desechadas durante el proceso evolutivo. En otras palabras, las mutaciones en los individuos pueden ser importantes en el proceso evolutivo si se propagan a través de la descendencia y comienzan a tener un papel en las poblaciones de la especie; pero si el individuo no se reproduce, la mutación, cualquiera que haya sido su efecto, se pierde evolutivamente. Un ejemplo de esto es la domesticación y evolución del maíz (Zea mays) desde su ancestro silvestre, el teocinte (Zea mays ssp. parviglumis), hace 10,000 años. Al igual que sucede de manera natural, durante la domesticación y el origen del maíz hubo una presión de selección hacia plantas que tuviesen una dominancia apical de un solo tallo, como sucede en el maíz, y no múltiples tallos de tipo arbustivo, como sucede en el teocinte. Lo que hoy sabemos es que el proceso evolutivo de domesticación favoreció que ciertas mutaciones ocasionadas por inserciones de transposones en un gen llamado teocinte branched 1 (tb1) fueran seleccionadas a tal grado que las variedades modernas de maíz no tienen el gen sin la mutación del transposón en tb1 (Hubbard et al., 2002) (Fig. 4B). Si las mutaciones en tb1 se hubieran dado en un individuo que no dejó descendencia y que se haya propagado en la población de teocinte, probablemente hoy no tendríamos tortillas ni tamales. Otro ejemplo que fue recientemente descubierto en el Instituto de Biología es la evolución de dos especies de abejas muy cercanamente relacionadas, que comparten distribución geográfica, pero que se diferencian en regiones del genoma que contienen genes de receptores de olores, lo cual les permite a ambas especies colectar diferentes aromas de las flores y utilizarlos en la reproducción sexual de manera específica y evitar el entrecruzamiento de las especies. Es decir, aunque en la mayor parte de los genomas de estas abejas es casi idéntico, hay selección de algunas mutaciones en ciertas regiones enriquecidas en genes receptores de olores, en lo que se llama islas de diferenciación genética. Si el grupo de abejas que tuvo las primeras mutaciones en los receptores de olor no se hubiese reproducido por ser más exitosas en su especificidad de apareamiento, probablemente no estaríamos hablando de dos especies de abeja, sino de una sola.
Referencias
CA, N. (1958). Studies on the Cytology and Phylogeny of the Pteridophytes VI. Observations on the Ophioglossaceae. Cytologia, 23(3), 291-316.
Clark, J. I., & Coen, E. S. (2002). The cycloidea gene can respond to a common dorsoventral prepattern in Antirrhinum. The Plant Journal, 30(6), 639-648.
Cui, L., Veeraraghavan, N., Richter, A., Wall, K., Jansen, R. K., Leebens-Mack, J., ... & depamphilis, C. W. (2006). ChloroplastDB: the chloroplast genome database. Nucleic acids research, 34(suppl_1), D692-D696.
Erickson, K. A., & Franciszkowicz, M. J. (2010). Problems with DNA. PRIMUS, 20(2), 140-147.
Formaggioni, A., Luchetti, A., & Plazzi, F. (2021). Mitochondrial genomic landscape: a portrait of the mitochondrial genome 40 years after the first complete sequence. Life, 11(7), 663.
Gualberto, J. M., Mileshina, D., Wallet, C., Niazi, A. K., Weber-Lotfi, F., & Dietrich, A. (2014). The plant mitochondrial genome: dynamics and maintenance. Biochimie, 100, 107-120.
Haberer, G., Young, S., Bharti, A. K., Gundlach, H., Raymond, C., Fuks, G., ... & Messing, J. (2005). Structure and architecture of the maize genome. Plant physiology, 139(4), 1612-1624.
Hayes, B. (1998). Computing science: The invention of the genetic code. American Scientist, 86(1), 8-14.
https://www.ncbi.nlm.nih.gov/refseq/annotation_euk/Physcomitrella_patens/100/
Hubbard, L., McSteen, P., Doebley, J., & Hake, S. (2002). Expression patterns and mutant phenotype of teosinte branched1 correlate with growth suppression in maize and teosinte. Genetics, 162(4), 1927-1935.
Liu, Q., Ding, C., Chu, Y. et al. PoplarGene: poplar gene network and resource for mining functional information for genes from woody plants. Sci Rep 6, 31356 (2016).
Lledías, F., Gutiérrez, J., Martínez-Hernández, A., García-Mendoza, A., Sosa, E., Hernández-Bermúdez, F., ... & Nieto-Sotelo, J. (2020). Mayahuelin, a type I Ribosome Inactivating Protein: characterization, evolution, and utilization in phylogenetic analyses of Agave. Frontiers in Plant Science, 11, 573.
McClintock, B. (1948). Mutable loci in maize. Carnegie Inst Wash Year Book, 47, 155-169.
National Library of Medicine. Genetics Home Reference. How can gene mutations affect health and development? www.ghr.nlm.nih.gov/handbook/mutationsanddisorders/mutationscausedisease
Palomino, G., Martínez-Ramón, J., Cepeda-Cornejo, V., Ladd-Otero, M., Romero, P., & Reyes-Santiago, J. (2021). Chromosome number, ploidy level, and nuclear DNA content in 23 species of Echeveria (Crassulaceae). Genes, 12(12), 1950.
Purugganan, M. D., & Jackson, S. A. (2021). Advancing crop genomics from lab to field. Nature genetics, 53(5), 595-601.
Pellicer J, Fay MF, Leitch IJ: The largest eukaryotic genome of them all?. Bot J Linn Soc. 2010, 164: 10-15. 10.1111/j.1095-8339.2010.01072.x.
*Foto de portada creada con Chat GPT
Ulises Rosas
Investigador Titular A del Jardín Botánico del Instituto de Biología de la UNAM. Obtuvo su Doctorado en el John Innes Centre del Reino Unido, y realizó un posdoctorado en el Center for Genomics and Systems Biology de la Universidad de Nueva York. Desde 2015, fundó una nueva rama de investigación en la UNAM con el fin de entender las bases genéticas y genómicas de la diversidad, principalmente en las raíces de cactáceas.
Brenda Anabel López Ruiz
Investigadora posdoctoral del Jardín Botánico de la UNAM. Está interesada en estudiar la biología del desarrollo de cactáceas y de suculentas mexicanas. Doctora en Ciencias Bioquímicas por el Departamento de Bioquímica, UNAM. M. en C. en la especialidad de Biotecnología por el CINVESTAV y Bióloga egresada de la FES-Iztacala, UNAM.